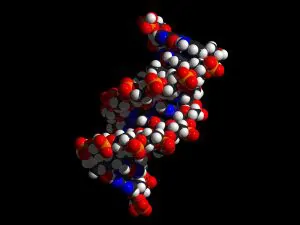
Naukowcy kodują całe bloki danych w DNA. Ta technologia jest przyszłościowa ze względu na możliwość zapisania całej zawartej w sieci informacji w urządzeniu wielkości kciuka.
Eksperyment
W 2012 roku naukowcy z Harwardu ogłosili, że opracowali możliwość zapisu i odczytu informacji z genów DNA. Dane zapisywane są w postaci binarnej, zero-jedynkowej. Zakodowano bity na parach DNA przypisując wartość zero dla nukleotydów A, C oraz wartość jeden dla G oraz T. Gęstość zapisu pozwoliła na sześcianie o boku 1 mm zapisać 5.5 petabajtów danych, czyli 1 milion GB danych. W związku z tym fiolka płynu może zawierać miliardy książek w postaci elektronicznej. Tego typu rozwiązanie nie jest narażone na typowe problemy przechowywania danych na dyskach magnetycznych i optycznych. Łańcuchy DNA są długowieczne a co za tym idzie informacje w nich zapisane również. Jednakże dane nie zostały zapisane w żywej komórce ze względu na marnowanie przestrzeni oraz groźbę zmutowania łańcucha i zmiany informacji. Prace nad podobnymi projektami prowadzone były równolegle w kilku ośrodkach naukowych. Pierwsza syntetyczna komórka stworzona w 2010 roku zawierała informację o nazwiskach naukowców realizujących projekt, ale dopiero eksperyment realizowany na Harwardzie zapisał znaczną ilość danych w postaci 53426 słów, 11 ilustracji i programu napisanego w JavaScript. Ilość zapisanych danych odpowiadała dyskietce 3.5 cala, czyli wynosiła około 1.4MB.
Archiwizacja danych
Ciekawym następstwem technologii jest możliwość archiwizacji danych za pomocą dwóch współpracujących bakterii. Aby tego dokonać pierwszą bakterię Novablue umieszcza się w obszarze wprowadzania danych, tak aby nie mogła się wydostać. W praktyce jest to powierzchnia, na której bakteria ma ograniczoną ruchliwość, np zawierającą streptomycynę. Następnie wprowadza się drugą bakterię typu HB101, której możliwości mobilne w środowisku są znacznie większe. Cały rejon otoczony jest substancją, która zapobiega ucieczce obu bakterii. Następnie dane przekazywane są pomiędzy bakteriami. Proces trwa około 72 godzin. Takie środowisko jest skalowalne, czyli można umieścić wiele bakterii przekazujących sobie informacje. Pozostaje problem, które bakterie powinny być sparowane, ale ten problem próbuje się rozwiązać za pomocą tworzenia śladów chemicznych, za którymi podążają bakterie, więc proces może być całkowicie pod kontrolą. Stąd następnym krokiem będzie stworzenie „nawigacji” dla bakterii, dzięki temu dane zapisane w DNA będzie można przekazywać pomiędzy bakteriami tworząc w zasadzie nieograniczoną ilość kopii bezpieczeństwa.